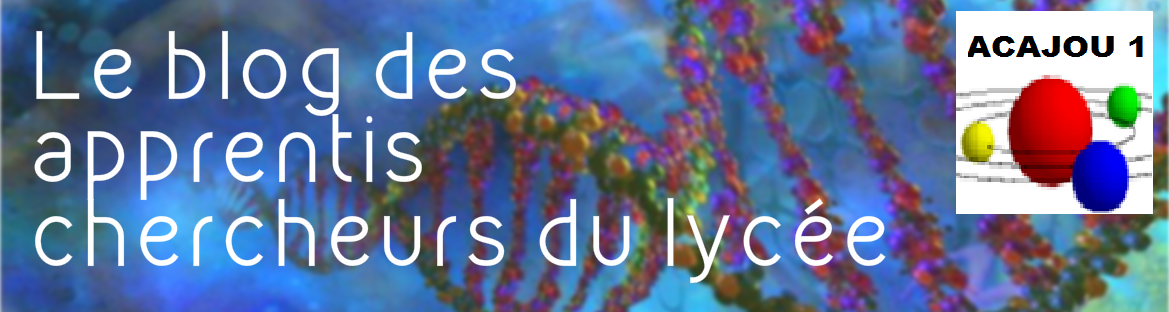LA DIVERSITE DES IGNAMES
Extraction d'ADN 2016-2017
L'ADN a été extrait des feuilles d'Ignames de 5 variétés : Poule, Pyramide, Portugaise, Belep et Cousse-Couche. Pour cela, nous avons utilisé différents matériels :
Un petit morceau de feuille d'Igname est découpé aux ciseaux. A l'aide du kit d'extraction QIAGEN, l'ADN sera purifié par divers traitements :
- des tampons qui vont neutraliser les membranes cellulaires
- deux mini-colonnes : une va retenir les débris cellulaires, la deuxième va retenir l'ADN purifié
- diverses centrifugations pour séparer les différents éléments cellulaires de l'ADN
Malheureusement, tout se fait "à l'aveugle" : on ne sait jamais si on a réussi à extraire l'ADN ou pas!! (car il est non visible tout au long du processus).
Amplification de l'ADN par PCR
Après avoir extrait l’ADN des feuilles d’ignames, il est nécessaire d’amplifier cet ADN car il est en trop faible concentration. Nous n’allons pas amplifier tout le génome d’Igname sur toute sa longueur, mais seulement certaines zones ciblées, grâce à une technique : la PCR (Polymerase Chain Reaction). Cette technique consiste à obtenir un très grand nombre de copies d’une zone cible d’ADN. Les copies sont fabriquées par une enzyme : l’ADN polymérase. Les zones à cibler sont repérées par des « amorces » :
Schéma du principe d’amplification par PCR
Nous avons mis dans chaque tube : l’extrait d’ADN d’Igname, une amorce, des nucléotides et des sels magnésium, et l’ADN polymérase. Puis, nous avons placé tous les tubes dans le thermocycleur programmé pour réaliser 35 cycles composés :
- d’une dénaturation (ouverture de la molécule d’ADN par chauffage à 95°C),
- d’une hybridation (accrochage de l’amorce à 49°C, 55°C ou 57°C selon l’amorce)
- et d’une élongation (fabrication d’ADN par l’ADN polymérase à 72°C).
La PCR a été réalisée avec huit amorces différentes, sur 2 extraits d’ADN de chacune des 5 variétés d’Igname de notre étude.
A chaque fois, un témoin négatif a été prévu (de l’eau à la place de l’extrait d’ADN dans le tube de PCR). Ainsi qu’un témoin positif (ADN de l’Igname INRAX17, déjà testé l’année passée).
Vérification des résultats
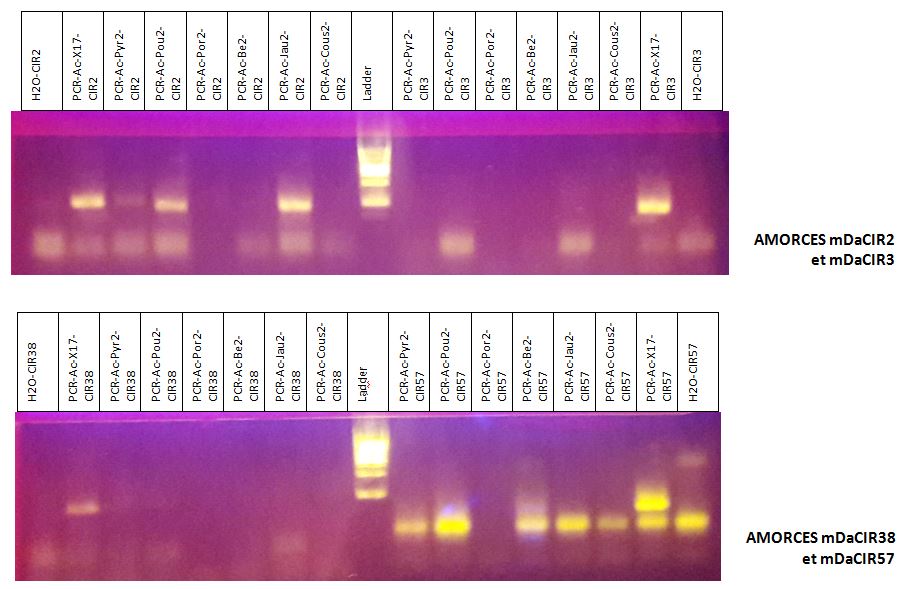
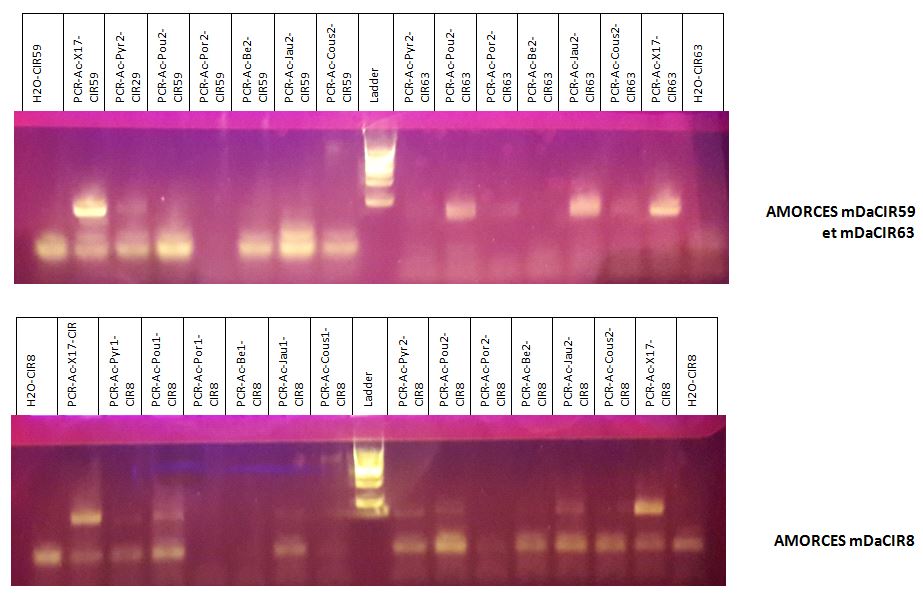
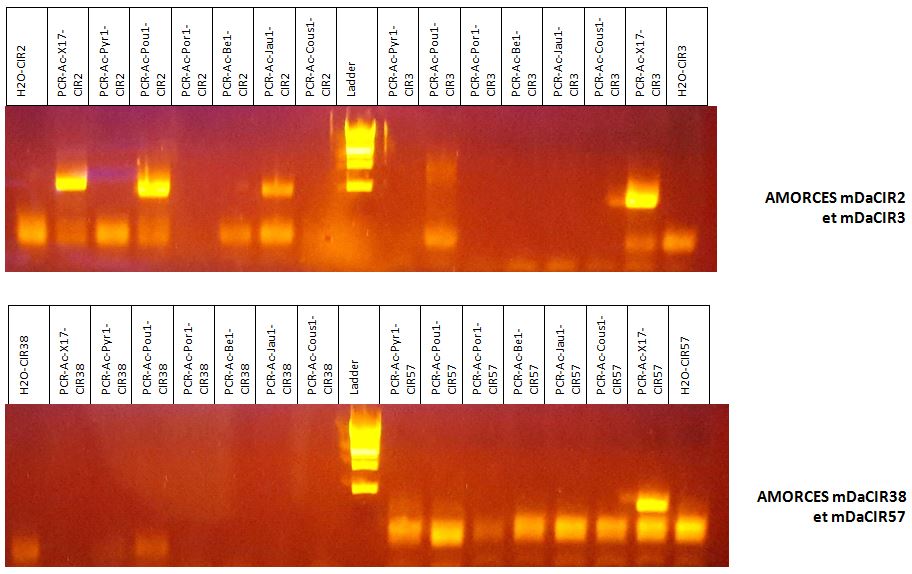
Après avoir réalisé l’amplification de l’ADN des différentes zones sur le génome par la PCR, nous avons effectué des tests sur mini-gels d’électrophorèse LONZA afin de vérifier si l’extraction et l’amplification de l’ADN ont bien été menées.

Chaque échantillon de PCR (5 µl) est déposé dans un puits du gel d'agarose :
Une fois la migration réalisée, on peut ensuite visualiser les bandes d'ADN en éclairant aux UV.
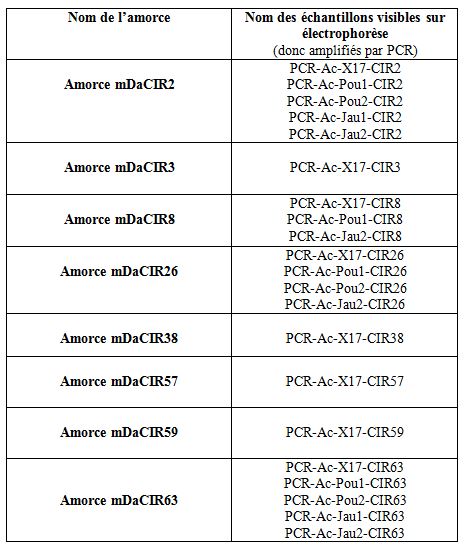
Les résultats sont les suivants :
mDaCIR2, mDaCIR3, ....mDaCIR63 sont les noms des 8 amorces utilisées pour cibler les zones à amplifier par PCR.
Le ladder est un marqueur de taille.
La présence des témoins négatifs (avec H2O) permet d'éliminer de la lecture des gels les bandes qui sont présentes tout en bas, puisqu'elles sont aussi présentes dans les témoins négatifs.
L'analyse des gels permet de retenir les résultats suivants :
Finalement, seules 3 variétés d'Igname seront retenues (INRAX17, Poule et Jaune) et 4 zones amplifiées (avec mDaCIR2, mDaCIR8, mDaCIR26 et mDaCIR63).
Ces échantillons peuvent être envoyés au Génoscope pour y être séquencés!! .....et c'est chose faite !!
Rendez-vous à la rentrée prochaine pour l'analyse des séquences....et la réponse à notre question......