tests génétiques
Extractions d'ADN
extraction d'ADNL'ADN de nos 5 variétés d'Igname (Pakala, Manlankon, Kabusah, INRAX17 et INRAX154) est extrait à partir de feuilles que Mme DUQUESNAY, notre enseignante, a récupéré, au mois de juin 2015 (quand les feuilles sont jeunes) sur la parcelle expérimentale de la Chambre d'Agriculture de Martinique, au quartier Chopotte au François (un grand merci à Mme JEAN-BAPTISTE pour son accueil et son soutien :)). Ces feuilles ont été congelées jusqu'à ce que nous les utilisions aujourd'hui.
C'est à partir d'un tout petit morceau de feuille que l'ADN va être extrait, grâce à un kit d'extraction "DNeasy Plant Mini Kit" de la société Qiagen. Cela consiste en plusieurs étapes successives, au cours desquelles la feuille va être broyée, les cellules et les membranes cassées, les différents débris cellulaires séparés de l'ADN par précipitations/centrifugations.
A la fin, on obtient à peu près 100 microlitres de liquide renfermant, on l'espère, l'ADN d'igname. Malheureusement, à ce stade, on n'est sûr de rien!! L'ADN est invisible!! On ne peut absolument pas savoir si tout notre travail a été correct!!
Qu'est ce que la PCR?
La PCR
PCR est l'abbréviation de l’expression anglaise Polymerase Chain Reaction ou Réaction en Chaîne par Polymérase (le terme français équivalent, Amplification en Chaîne par Polymérisation ou ACP, est rarement utilisé) est une technique de réplication ciblée in vitro.
Elle permet d'obtenir, à partir d'un échantillon complexe et peu abondant, d'importantes quantités d'un fragment d'ADN spécifique et de longueur définie. L'ordre de grandeur à retenir est celui du million de copies en quelques heures(on dit que l'amplification est exponentielle).
Le principe et les conditions expérimentales qui en découlent sont très simples. Il s'agit de réaliser une succession de réactions de réplication d'une matrice double brin d'ADN. Chaque réaction met en oeuvre deux amorces oligonucléotidiques dont les extrémités 3-prime pointent l'une vers l'autre. Les amorces ou «primers» en anglais définissent alors, en la bornant, la séquence à amplifier.
L'astuce consiste à utiliser les produits de chaque étape de synthèse comme matrices pour les étapes suivantes, au lieu de les séparer afin de ne réutiliser que la matrice originale.
Le principe de la PCR est expliquée en vidéo en suivant ce lien : vidéo expliquant la PCR
Conditions de nos PCR
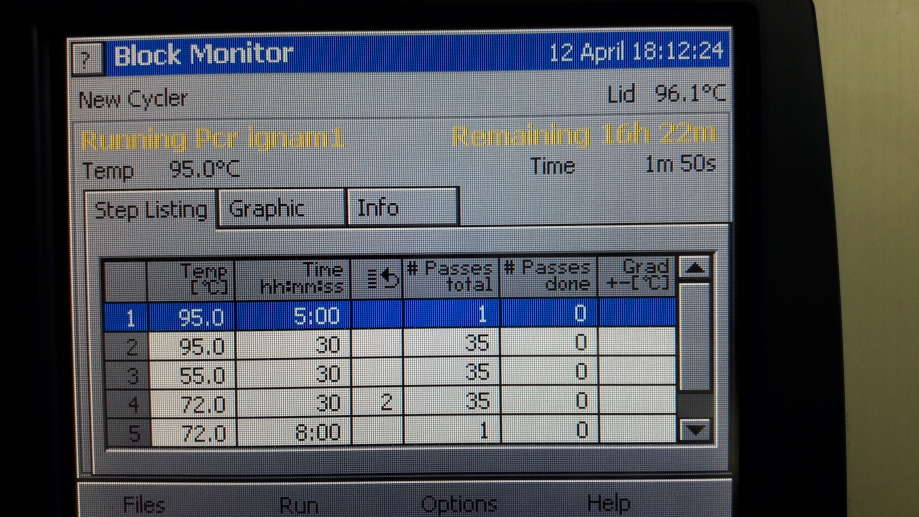
Nous avons réalisé nos PCR grâce à un appareil appelé thermocycleur
Chaque tube renferme :
- 2 microlitres d'ADN (ou d'H2O pour le témoin négatif)
- 13 microlitres de mix H2O + amorces (par couple pour "borner" la zone à amplifier)
- 15 microlitres d'enzyme Taq polymérase
Nous avons réalisé des PCR sur des extractions d'ADN de divers échantillons (=feuilles différentes) des variétés d'Ignames (ex : Pakala-1 = échantillon 1 / Pakala-3 = échantillon 3).
Nous avons réalisé ces PCR avec 6 couples d'amorces, notées mDaCIR2, mDaCIR3, mDaCIR38, mDaCIR57, mDaCIR59 et mDaCIR63. Les séquences de ces amorces ont été fournies par Mme Dalila PETRO de l'INRA. Elles ont été fabriquées par l'entreprise Eurogentec, puis nous ont été envoyées par l'Ecole de l'ADN de Nîmes (tout ceci gracieusement!!)
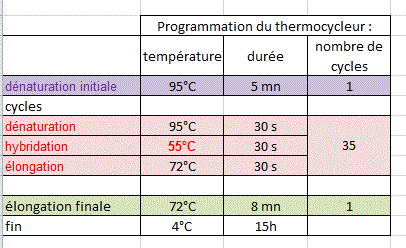
Les cycles programmés pour ces PCR furent les suivants :
Le thermocycleur gère seul les changements de température et la répétition des cycles.
A la fin, les tubes sont maintenus à 4°C jusqu'à ce qu'on les récupère. On les place alors au congélateur jusqu'au jour où on peut tester le résultat sur électrophorèse.
Électrophorèse: principe de l'électrophorèse
L'électrophorèse sur gel d'agarose est une méthode utilisée en biochimie et en biologie moléculaire pour séparer l'ADN, l'ARN ou des protéines en fonction de leur poids moléculaire.
La technique de l'électrophorèse sur gel d'agarose est basée sur la séparation des acides nucléiques chargés négativement sous l'effet d'un champ électrique. Cette séparation s'effectue à travers la matrice du gel d'agarose : les molécules de plus petites tailles se déplacent plus rapidement et migreront plus loin que les molécules de tailles supérieures.
L'électrophorèse consiste à faire migrer des ions sous l'effet d'un courant électrique égal à 275 V. Chaque échantillon est déposé dans un puits du gel d'Agarose.
La méthode de révélation la plus utilisée est la révélation au bromure d'éthydium ou BET. Le bromure d'éthydium est un agent d'intercalation couramment utilisé comme marqueur d'acide nucléique dans les laboratoires de biologie moléculaire. Lorsqu'il est exposé à des rayonnements ultraviolets, il devient fluorescent avec une couleur rouge-orangée, 20 fois plus intense lorsqu'il est lié à l'ADN.
Le bromure d'éthydium étant un produit dangereux , nous ne l'avons pas utilisé.
Nos électrophorèses ont été réalisées sur des gels prêts à l'emploi de la marque LONZA (système FlashGel pour ADN concentré à 2,2%). Les bandes d'ADN sont révélées par une lampe UV intégrée dans le support du gel.
Résultats de nos électrophorèses
Les échantillons amplifiés par PCR sont soumis à électrophorèse sur les flashgel LONZA.
- H2O = témoin négatif (aucun ADN dans le tube sauf les amorces)
- marqueur de taille = sert de référence de tailles des bandes d'ADN / n'a pas subi de PCR
- pakala-1 = échantillon (feuille) n°1 de la variété Pakala
- témoin tolérant = PCR réalisée avec l'ADN d'une variété d'Igname connue pour être tolérante à l'anthracnos
- témoin sensitif = PCR réalisée avec l'ADN d'une variété d'Igname connue pour être sensible à l'anthracnose
Le microsatellite mDaCIR3 varie, selon les variétés, entre 204 et 228 paires de bases (données de D. PETRO). On constate une même bande, pouvant correspondre à cette taille, pour le témoin tolérant et l'échantillon INRAX17 n°3. Par contre, on voit une contamination du puits Pakala-1 par le marqueur de taille.

Le microsatellite mDaCIR38 varie, selon les variétés, entre 218 et 241 paires de bases (données de D. PETRO). On constate comme précédemment une même bande pour le témoin tolérant et INRAX17, à une taille proche de 250 pb.

Le microsatellite mDaCIR57 varie, selon les variétés, entre 165 et 207 paires de bases (données de D. PETRO). On aperçoit ici des bandes dans les puits de Pakakla-1, Kabusah-2, INRAX154-1, Kabusah-3 (faible) et INRAX17-3 qui sont toutes à la même hauteur. Le problème c'est qu'on voit la même bande dans le puits du témoin négatif H2O, sans ADN! On ne peut donc pas en tenir compte. Par contre, on voit encore la même bande, en peu plus haute, pour le témoin tolérant et INRAX17 n°3.
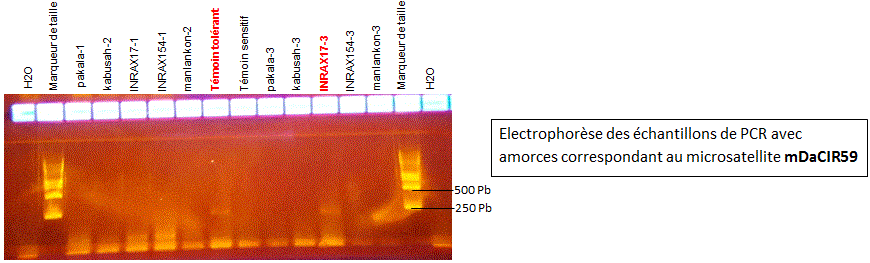
Le microsatellite mDaCIR59 varie, selon les variétés, entre 210 et 237 paires de bases (données de D. PETRO). Comme précédemment, on voit des bandes en bas du gel présentes dans tous les puits, même les témoins négatifs sans ADN (H2O) : on n'en tient pas compte. Il y a encore deux bandes identiques, mais faibles, pour le témoin tolérant et INRAX17 n°3.
Les gels d'électrophorèse des microsatellites mDaCIR2 et mDaCIR63 ne sont malheureusement pas exploitables.
En conclusion, l'échantillon n°3 de la variété INRAX17 a à chaque fois une bande identique au témoin tolérant. On peut en déduire que la variété INRAX17 est tolérante à l'anthracnose.